Разработка информационной модели системы прогнозирования многофакторных заболеваний
Моделирование предметной области
Анализ эффективности модели БД
Подавляющее большинство наследственных болезней является многофакторными, то есть обусловленными как наследственными факторами, так и, в значительной мере, воздействием окружающей среды. Комплексный подход к выявлению всех факторов риска возникновения заболевания позволяет получить адекватную вероятностную модель его развития.
В настоящее время существует ряд программных средств, позволяющих проводить диагностику предрасположенности пациента к заболеваниям. Однако эти средства учитывают только генетическую составляющую. Основным преимуществом разрабатываемой системы является учет особенностей влияния внешней среды при прогнозировании наследственных патологий. В работе представлена информационная модель системы прогнозирования многофакторных заболеваний. Разработана структура представления данных, позволяющая смоделировать имеющиеся в предметной области отношения.
Многофакторные заболевания
Информация о структуре человеческого организма содержится в молекуле ДНК. За формирование каждого признака отвечает ген, локализованный на некотором участке ДНК. Аллель — состояние гена, обусловленное последовательностью нуклеотидов; именно аллель определяет индивидуальное проявление признака. Анализируя состояние гена, мы сталкиваемся с генетическими модификациями, которые не только влекут за собой вариацию нормальных признаков, но и могут стать причинами болезней. До последнего времени внимание в наследственной патологии акцентировалось на моногенных болезнях, возникающих в результате повреждения ДНК на уровне отдельного гена. С развитием методов анализа генома внимание исследователей сфокусировалось на многофакторных болезнях. Эта группа болезней в настоящее время составляет 92% от общего числа наследственных патологий человека. Многофакторные болезни обусловлены как наследственными факторами, так и, в значительной мере, факторами воздействия внешней среды. Кроме того, они связаны с действием многих генов, поэтому их также называют полигенными. В отличие от моногенных заболеваний, не зависящих от влияния внешней среды и наследуемых в соответствии с законами классической генетики, когда 25 или 50% родственников больного первой степени также больны, многофакторные болезни обычно выявляются у 510% таких родственников.
В процессе синтеза дочерней молекулы ДНК могут происходить ошибки. Один нуклеотид может быть пропущен или заменен другим. В некоторых случаях целый участок ДНК может быть удален, удвоен или развернут. Такие события называются мутациями (полиморфизмами). За одно поколение человечество накапливает множество мутаций, при этом большинство полиморфизмов не оказывают влияния на человеческий организм.
Влияние наследственных патологий на заболевание осуществляется в тесном взаимодействии с внешней средой. Можно сказать, что фенотипические признаки — это факторы окружающей среды. В качестве таких факторов могут выступать климат, экология, питание, особенности жизнедеятельности и поведения человека. Таким образом, каждый организм характеризуется набором генетических и фенотипических признаков.
При построении системы прогнозирования многофакторных заболеваний используется комплексный подход к определению всех факторов риска возникновения заболевания. В предметной области можно выделить следующие основные классы:
- ген (Gene);
- полиморфизм (SNP);
- заболевание (Disease);
- фенотипический (средовый) признак (Phenotype);
- медицинское исследование, позволяющее определить значение шанса развития исхода (OR).
Медицинское исследование можно представить как описание заболевания и набор факторов риска, имеющийся у изучаемой группы. Для каждого исследования обычно приводится значение величины OR. OR определяется как отношение шанса развития исхода при воздействии фактора риска к шансу развития исхода без воздействия фактора риска. OR не является оценкой вероятности и поэтому может принимать значения больше 1.
В рассмотренной модели существуют две «многозначные» зависимости: «Заболевание» — «Фенотип» и «Заболевание» — «Полиморфизм», то есть заболевание может возникнуть при условии существования сразу нескольких, причем, возможно, разных факторов риска. То есть при описании предметной области возникают кортежи неопределенной длины вида:
(<З1>, <Ф1>,…, <Фn>, <П1>, …, <Пm>).
Для их описания не существует адекватных средств стандартного UML, и их, в принципе, нельзя представить с помощью реляционной модели.
Новый тип отношений
Перед нами новый тип отношений, которые могут возникать между классами, точнее между объектами классов. С одной стороны в отношении нового типа участвует один объект одного класса, а с другой — произвольное число объектов второго класса. То есть отношение задается набором кортежей переменной длины.
Перед тем как классифицировать новое отношение как разновидность отношения ассоциации, остановимся подробнее на уже известном нам отношении, существующем между классами «Заболевание» и «Полиморфизм». Отношения между объектами этих классов можно кратко описать так: объект «Заболевание» может быть связан с любым количеством объектов «Полиморфизм», причем кортежи разной длины характеризуются еще и дополнительным атрибутом — вероятностью заболевания при условии наличия указанных генотипических признаков.
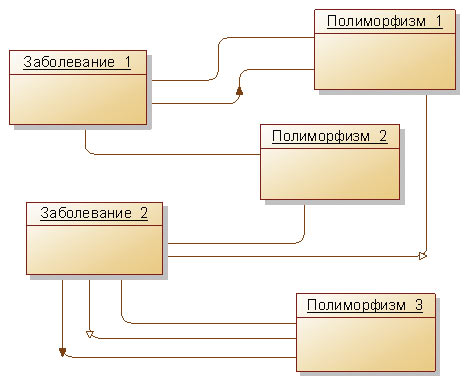
Рис. 1. Диаграмма объектов
Если отображать связи между объектами графически, то получится, что один объект класса «Заболевание» может быть связан с одним объектом «Полиморфизм» более одного раза. Для того чтобы различать разные отношения, существующие между одними и теми же объектами разных классов, они должны быть поразному помечены. То есть диаграмма объектов при наличии описываемых отношений может иметь такой вид, как на рис. 1. Этот рисунок задает следующие кортежи — отношения между объектами:
(З1, П1), (З1, П1, П2), (З2, П2, П3), (З2, П3), (З2, П1, П3).
Очевидно, что средств для описания и задания такого рода отношений в языке UML нет. Но в UML есть механизм стереотипов, который позволяет расширять стандартный набор строительных блоков языка. В данном случае расширить надо отношение ассоциации, то есть, как уже было сказано, ввести новый тип отношения ассоциации.
Следует отметить, что приведенные примеры отношений нового типа не относятся к одной конкретной предметной области, а носят универсальный характер. Для нового отношения предлагается ввести название «вариативная ассоциация» и новое графическое изображение (рис. 2).

Рис. 2. Новый вид отношений
Свойства новой ассоциации совпадают со свойствами обычной ассоциации. Имя и роли не требуют пояснений. Кратность задается так же, как и для обычной ассоциации, но со стороны класса, делегирующего в ассоциацию несколько экземпляров объектов, следует задавать два показателя кратности, причем второй из них задает число объектов класса, участвующих в конкретной ассоциации.
Например, (*, 1..4).
Чаще всего с вариативной ассоциацией связаны дополнительные атрибуты, что делает необходимым введение по аналогии с классомассоциацией класса «вариативная ассоциация». Такой класс может изображаться точно так же, как и обычный классассоциация.
Моделирование предметной области
Модель предметной области, представленная в виде диаграммы классов, будет иметь такой вид, как на рис. 3.
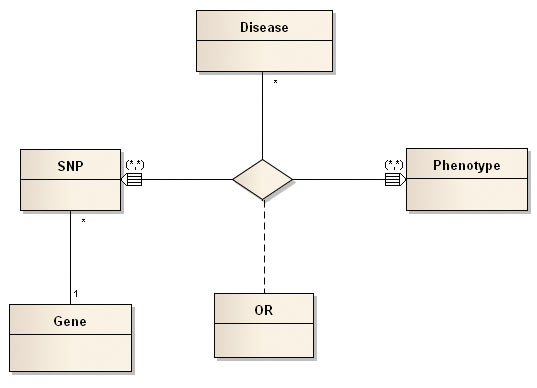
Рис. 3. Диаграмма классов предметной области
Отметим, что между классами «Заболевание», «Полиморфизм» и «Фенотипический признак» существует тернарная ассоциация. Отношение обычной ассоциации между классами «Полиморфизм» и «Ген» имеет кратность 1 и *, поскольку в одном гене может присутствовать сразу несколько полиморфизмов, а полиморфизм принадлежит строго определенному гену.
Тем не менее для реализации нашей информационной системы будет использоваться реляционная СУБД. В связи с этим необходимо представлять в виде стандартных реляционных отношений не только отношения «многие ко многим», но и вариативные ассоциации.
Рассмотрим вопрос представления, точнее моделирования отношения вариативной ассоциации. Сделаем это на примере отношения между классами «Заболевание» и «Полиморфизм». Введем дополнительный класс «Группа_полиморфизмов». Этот класс свяжем отношением «один ко многим» с классом «Полиморфизм» и отношением «многие ко многим» с классом «Заболевание».
Тогда объект класса «Заболевание» через объект класса «Группа_полиморфизмов» будет связан с произвольным числом объектов класса «Полиморфизм». То есть каждое конкретное заболевание косвенно оказывается связанным с несколькими наборами полиморфизмов. Другими словами, кортеж (З2, П1, П2, П3) представляется с помощью четырех кортежей: (З2, ГП1), (ГП1, П1), (ГП1, П2) и (ГП1, П3), а кортеж (З2, П1, П4) — с помощью трех кортежей: (З2, ГП2), (ГП2, П1) и (ГП2, П4).
Всё то же самое касается и отношения, существующего между классами «Заболевание» и «Фенотипический признак». Для его представления введем дополнительно еще один класс «Группа_фенотипических_признаков» и точно так же свяжем его отношением «один ко многим» с классом «Фенотипический признак» и отношением «многие ко многим» с классом «Заболевание».
В результате всех этих действий два отношения вариативной ассоциации оказываются упраздненными. Но остается еще тернарная ассоциация, которая возникнет между классами «Заболевание», «Группа_полиморфизмов» и «Группа_фенотипических_признаков». Ассоциативный класс OR обеспечит поддержку свойств этой тернарной ассоциации. Кроме того, введение этого класса даст возможность избавиться от отношений «многие ко многим», существующих между классами «Заболевание» и «Группа_полиморфизмов» и «Заболевание» и «Группа_фенотипических_признаков», и заменить их отношениями «один ко многим».
Полученная логическая схема базы данных представлена на рис. 4.
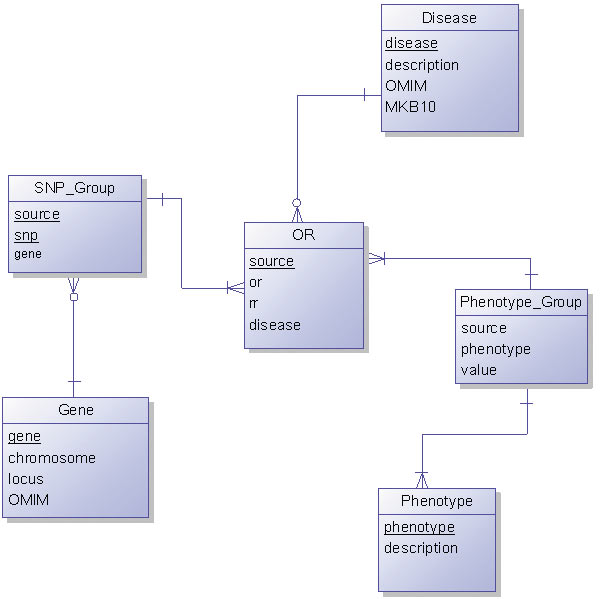
Рис. 4. Логическая схема базы данных
Для связи факторов риска и значений шансов возникновения заболевания используются таблицы связи OR_SNP и OR_Ph. Каждому исследованию соответствует уникальный индекс, с помощью которого выражается связь медицинских исследований и полиморфизмов в таблице OR_SNP. Этот индекс также осуществляет связь с фенотипическими признаками в таблице OR_Ph. Таким образом, мутации и фенотипические признаки связаны между собой через OR.
Выделенные сущности будут иметь следующие атрибуты:
- Gene (gene — название гена; chromosome — хромосома, на которой расположен ген; locus — положение на хромосоме; OMIM — индекс в медицинской библиотеке);
- SNP (snp — название полиморфизма; gene — ссылка на ген; rs — индекс в библиотеке NCBI);
- Disease (disease — название; description — описание; OMIM – индекс в медицинской библиотеке; МКБ10 — индекс в международной статистической классификации болезней);
- Phenotype (phenotype — название фенотипического признака; description — описание);
- OR (source — источник медицинского исследования; or — отношение шансов развития заболевания; rr — относительный риск заболевания у контрольной группы);
- SNP_Group (source — ссылка на медицинское исследование; snp — ссылка на полиморфизм);
- Phenotype_Group (source — ссылка на медицинское исследование; phenotype — ссылка на фенотипический признак; value — степень воздействия фактора риска).
Реализация полученного решения не будет зависеть от числа полиморфизмов и фенотипических признаков: при добавлении новых факторов риска структура данных будет сохраняться.
Анализ эффективности модели БД
Проведем расчет количества записей и полей в построенной информационной модели. Пусть R — количество полей. Введем функцию L(t), которая определяет количество записей в таблице t.
Количество записей в таблице OR определяется числом различных сочетаний полиморфизмов и фенотипических признаков. Пусть на каждое заболевание оказывает воздействие sor полиморфизмов и por фенотипических признаков. Всего различных сочетаний полиморфизмов и факторов риска окружающей среды для некоторого заболевания будет:
ASNP OR= Sor!
APh OR= Por!
Если d — количество заболеваний, то:
L(OR)=ASNP OR •APh OR •d
Поскольку в таблице имеются четыре атрибута, получим следующее число полей:
ROR = 4•L(OR) = 4•(ASNP OR •APh OR •d)
Таблица SNP_Group реализует связь между полиморфизмами и медицинскими исследованиями. Количество записей этой таблицы определяется числом различных сочетаний полиморфизмов для каждого заболевания:
L(SPNOR ) = ASNP OR •d
Учитывая, что в таблице имеются два атрибута, суммарное количество полей будет определяться следующим образом:
RSNP OR = 2•ASNP OR •d
Аналогично, таблица Ph_Group реализует связь между фенотипическими признаками и медицинскими исследованиями. Здесь число записей будет складываться из числа различных сочетаний фенотипических признаков по каждому заболеванию. Число полей этой таблицы будет определяться так:
RPhOR = 2•(APhOR •d)
Таблица Phenotype имеет два атрибута и содержит p записей, по числу различных фенотипических признаков. Следовательно:
RPhenotype = 2•p
Тогда вся представленная модель будет иметь следующее количество полей:
R = ROR + RSNP OR + RPh OR + RPhenotype + RDisease + RGene =
= 4•(ASNP OR•APh OR •d) + 2•ASNP OR •d + 3•s + 2 (APh OR•d) +
+ 2•p + 4•d + 4•g
Используя усредненные данные по медицинским исследованиям, проведем расчет объема занимаемой памяти. В качестве исходных данных возьмем следующие значения:
- количество заболеваний: d = 102;
- количество полиморфизмов: s = 4•102;
- количество фенотипических признаков: p = 102;
- количество генов: g = 5•102;
- будем считать, что на каждое заболевание оказывают воздействие в среднем Sor= 3 полиморфизма и Por = 5 фенотипических признаков.
Эти данные соответствуют значениям общего числа медицински значимых полиморфизмов и фенотипических признаков, учитываемых в ходе исследований. Подставив эти значения в формулу, получим
R = 330200
Учитывая, что каждое поле имеет размер 4 байта, получим объем памяти в 1,26 Мбайт.
Заключение
Статья посвящена проектированию информационной модели системы прогнозирования многофакторных заболеваний, в основе которой лежит статистическая модель развития патологии.
Реализация отношения между сущностями при образовании кортежей неопределенной длины не может быть выполнена с помощью стандартной реляционной модели. Для рассматриваемой предметной области это отношение возникает между сущностями «Заболевание», «Полиморфизм» и «Фенотипический признак». Была разработана структура представления данных, позволяющая смоделировать отношения, имеющиеся в предметной области. Произведен расчет объема памяти, необходимой для хранения данных.


